
Esercizi svolti in QGIS – Guida pratica all’analisi vettoriale
7 Settembre 2018
IIX Congresso Europeo di Mammalogia a Varsavia
5 Aprile 2019L’idea di scrivere questo tutorial nasce da un post facebook sul gruppo GIS ITALIA in cui si chiede come calcolare le aree di foraggiamento di una specie di chirotteri a partire da punti di presenza, utilizzando il Kernel Density. In biologia la stima di densità Kernel (Kernel Density Estimation KDE) è una tecnica utilizzata per il calcolo dell’home range delle specie (l’area in cui una specie vive e si muove) e fornisce una stima di densità dell’utilizzo del territorio. Il risultato dell’analisi KDE è una mappa raster che rappresenta la porzione di territorio utilizzata più frequentemente. Molto spesso i biologi sono interessati a due livelli di densità: il 95% e il 50% (core home range); il 95% rappresenta l’area nella quale la probabilità di trovare la specie è 0.95; il 50% rappresenta l’area nella quale questa probabilità è dello 0.5. Un altro metodo ancora utilizzato seppur abbastanza obsoleto è il Minimum Convex Polygon (MCP) che consiste semplicemente nel costruire, a partire da punti di presenza di una specie, il più piccolo poligono convesso intorno ai punti (molto spesso tende a sovrastimare il reale home range).
Un problema per nulla banale nel calcolo del KDE è la scelta del bandwidht o search radius. Il Bandiwdth controlla la “lisciatura” o smoothness ossia l’effetto dei punti sulla stima della densità. Esistono vari metodi che consentono di stimarlo partendo dai dati di presenza a disposizione:
- utilizzando alcune “regole” (ad esempio la funzione di Silverman, 1986);
- utilizzando algoritimi di cross-validazione;
- valutando la bontà dal punto di vista visivo.
Ad esempio ArcGIS utilizza una versione modificata della funzione di Silverman come parametro di default per il calcolo della KDE. Sfortunatamente in QGIS 3.4 non esiste una procedura automatica per la selezione dell’ottimo bandwidth (In QGIS 2.18 esiste il plugin AniMove che permette di selezionare diversi metodi). Tuttavia con un po’ di pazienza e codice è possibile implementare la funzione di Silverman in QGIS 3 ed ottenere un risultato molto soddisfacente (Alla fine della pagina c’è un link ad un progetto github dove abbiamo condiviso un modello di QGIS che automatizza completamente la procedura). La guida in linea di ArcGIS riporta la seguente funzione:
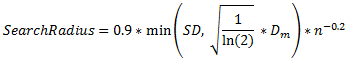
[eqn 1]
- min indica che il valore minimo tra i due indicati in parentesi deve essere utilizzato nell’equazione;
- SD è la Standard Distance
- Dm è la mediana delle distanze tra il centro medio dei punti di presenza e i punti stessi
- n è il numero di punti
La Standard Distance è data da:
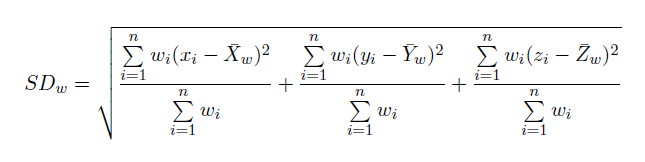
[eqn 2]
dove x, y sono le coordinate dei punti, X, Y sono le coordinate del centro medio dei punti, n rappresenta il numero dei punti. L’implementazione della funzione in QGIS è un po’ laboriosa e richiede l’utilizzo dal Calcolatore di campi (per una guida in italiano molto ben fatta del Calcolatore di Campi si può consultare la seguente guida).
Carichiamo i nostri punti di presenza della specie. Dobbiamo trovare il centro medio dei punti attraverso la funzione Media Coordinate (Vettore, Strumenti di Analisi).

In Layer in ingresso vanno caricati i punti di presenza e si può lasciare il resto di default. Il risultato è un punto (Coordinate medie) che rappresenta il centro medio dei nostri punti (il file è temporaneo ma è importante salvarlo cliccando con il tasto destro nel pannello dei layer e selezionare Make permanent).

A questo punto va calcolata la Standard Distance. Apriamo il Calcolatore di Campi del layer Punti di presenza e creiamo una nuovo colonna x_diff (Numero decimale, Lunghezza 10 e Precisione 3) che rappresenta la prima parte dell’equazione:
Nella finestra delle espressioni digitiamo:
($x-(x(geometry(get_feature_by_id('Coordinate medie', 0)))))^2
Importante: la funzione get_feature_by_id utilizza come argomenti il nome del layer sorgente da cui dobbiamo prendere la coordinata x e l’id del records che identifica quel punto. Se il file è temporaneo al posto dello 0 va indicato 1. Per i file salvati fisicamente (ad esempio tramite Make permanent) va indicato 0.
Stessa procedura per la creazione del campo y_diff
($y-(y(geometry(get_feature_by_id('Coordinate medie', 0)))))^2
Per il calcolo finale di SD bisogna, sempre dal Calcolatore di Campi, creare un nuovo campo sd (Numero decimale, Lunghezza 10 e Precisione 3) e digitare la seguente espressione:
sqrt((sum("x_diff")/ sum(@row_number) ) + (sum("y_diff")/sum(@row_number)))
Adesso calcoliamo il Dm (Median Distance). Apriamo il Calcolatore di Campi (sempre relativo al layer Punti di presenza) e aggiungiamo un nuovo campo dm (Numero decimale, Lunghezza 10 e Precisione 3) e inseriamo l’espressione:
median(distance($geometry, geometry(get_feature_by_id('Coordinate medie', 0))))
A questo punto abbiamo tutti gli elementi dell’equazione ma ci resta da determinare quale valore utilizzare (argomento min nell’eqn 1). Creiamo un nuovo campo che per comodità chiameremo ln (Numero decimale, Lunghezza 10 e Precisione 3) e inseriamo le seguente espressione:
sqrt( 1/ ln( 2) ) * "dm"
A questo punto possiamo valutare il valore minimo. Per calcolare il search radius creiamo un nuovo campo sr (Numero decimale, Lunghezza 10 e Precisione 3) e inseriamo le seguente espressione:
0.9*min( "sd" , "ln" ) *(sum(@row_number)^-0.2)
Se avete eseguito tutti i passaggi per bene, con i dati forniti nell’esempio, il valore del Search Radius è di 861.613 metri. Salviamo le modifiche apportate alla tabella attributi.
Negli strumenti di Processing cerchiamo l’algoritmo Mappa di concentrazione.

In Raggio andremo ad inserire il valore calcolato con il metodo di Silverman. La dimensione del pixel va scelta in base alle nostre esigenze (compromesso tra ampiezza dell’area studio, visualizzazione, pesantezza del raster di output): 50 metri sono sufficienti in questo caso. Premiamo su Esegui.

Il risultato è soddisfacente. Nello specifico è possibile distinguere tre areali più utilizzati dalla specie. Come detto all’inizio del tutorial, i biologi di solito calcolano due soglie di KDE: 50% e 95%. Si tratta di calcolare il 50° (la mediana) e il 95° percentile. Il prossimo passaggio è un po’ meccanico ma funzionale.
Per il calcolo della soglia al 50% (core home range) e della soglia al 95% bisogna eseguire i seguenti passaggi:
- tra gli strumenti di Processing selezionare Sample raster values: in Input Point Layer scegliere i Punti di presenza e in Raster Layer to sample la Heatmap (KDE) appena creata;
- aprire la tabella attributi del layer risultante dall’operazione 1) Sampled points;
- ordinare le righe della tabella in ordine decrescente in base al campo rvalue. Dividere il numero di punti (62 nell’esempio) per 0.5. Il valore è 31. Contare, partendo dall’alto, 31 righe e leggere in corrispondenza della 31 esima riga il valore rvalue. Il valore risultante è 3.7496;
- ripetere la stessa operazione moltiplicando questa volta 62 per 0.95 (59). Il valore risultante è 2.13391;

I due valori serviranno per riclassificare la Heatmap (KDE) in due classi. Per la core home range (50%) cerchiamo la funzione Riclassifica con tabella negli strumenti di processing e inseriamo i parametri come da immagine:

Salviamo il raster come core home range.tif. Per l’home range (95%) basta cambiare il valore minimo lasciando inalterati gli altri parametri. Salviamo il raster come home range.tif.
Ora è possibile vettorializzare i due raster con il comando Poligonizzazione (Raster, Conversione) e calcolare l’area dei due home range tramite il calcolatore campi con la funzione $area. Per migliorare la visualizzazione del vettore è possibile applicare algoritmi si lisciatura, ad esempio tramite il comando lisciatura nel menu Processing.

Il link al progetto github.
Per scaricare i dati di esempio del tutorial [download id=”10430″]


4 Comments
Ho provato ad utilizzare il modello su uno shp di 6 punti. Ho lasciato tutti i parametri di default, ma la procedura non va a termine (incollo sotto il log).
Utilizzo Win8, QGIS 3.4.3.
Probabilmente è un mio problema (forse non ho istallato qualcosa)…….
Grazie per un suggerimento su come posso risolvere il problema
Salve, probabilmente ha un problema con SAGA. Al momento il modello prevede l’utilizzo di algoritmi di QGIS e per un’operazione anche un algoritmo di SAGA (che nel suo caso fallisce). Abbiamo modificato il modello in modo da escludere SAGA e funzionare solo con QGIS
Grazie del tutorial, mi sorge il seguente dubbio: nell’ espressione 0.9*min( “dm” , “ln” ) *(sum(@row_number)^-0.2) non manca la deviazione standard?
Grazie per la segnalazione. In realtà SD è la Standard Distance. Nella formula è un refuso che abbiamo prontamente corretto (alla fine del risultato cambia davvero poco, si tratta di pochi metri di differenza). Provato ad utilizzare il modello di QGIS?